Algorytmy ewolucyjne i inteligentny projekt
Działający pod systemem operacyjnym Windows program Weasel (łasica) jest prostą prezentacją ewolucyjnego (genetycznego) algorytmu i służy do badania komputerowych symulacji ewolucyjnych procesów. Powstał on na bazie słynnej symulacji autorstwa znanego oksfordzkiego biologa i popularyzatora teorii ewolucji, Richarda Dawkinsa. W swojej książce Ślepy zegarmistrz opisał on taką symulację jako ilustrację potęgi doboru kumulatywnego. Program Weasel nie tylko dokładnie odwzorowuje symulację autorstwa Dawkinsa, ale posiada wiele dodatkowych opcji i funkcji, które wprowadzają do niej nieco biologicznego realizmu. Są to m.in. określanie tempa mutacji, ich rodzajów, liczby potomstwa itp. Program zawiera także opcję modelowania ewolucji projektowanej przez użytkownika sekwencji aminokwasów opartych na odpowiednich kodonach DNA. Program Weasel przez swoją prostotę, intuicyjność i wielofunkcyjność jest doskonałym narzędziem dla wszystkich zainteresowanych algorytmami genetycznymi — zarówno początkujących, jak i pasjonatów, w prosty sposób demonstrując fałszywość zapewnień, że ewolucyjne algorytmy potwierdzają teorię ewolucji.
POBIERZ PROGRAM WEASEL (895 KB ZIP)
Uwaga: Program jest prosty i intuicyjny, ale w wersji angielskiej. Dlatego mocno sugerujemy, aby pierwsze kroki z programem Weasel stawiać mając przed oczami Przewodnik po programie Weasel . Ułatwi on znacznie początkującemu użytkownikowi korzystanie z tego ewolucyjnego symulatora.
Algorytmy ewolucyjne zaczęły robić karierę z końcem lat 60-tych i początkiem 70-tych ubiegłego wieku, kiedy John Holland wraz ze swoimi studentami z University of Michigan stworzyli program zwany algorytmem genetycznym do symulowania procesu ewolucji w naturze. [1] Od tego czasu na tym polu nastąpił ogromny postęp; komputerowe symulacje oferują idealne warunki, dzięki który powolny i rozciągnięty w czasie proces ewolucji może zostać in silico przyspieszony. Wedle zapewnień ewolucjonistów, algorytmy genetyczne zawierają również podstawowe charakterystyki tego procesu. Otwiera to nowe możliwości pomocne w udzielaniu odpowiedzi na takie pytania, jak: w jaki sposób darwinowski mechanizm prowadził do powstawania nowych funkcji, do znajdowania szeregu rozwiązań spełniających cechy projektu? Jest rozpowszechnionym poglądem, że ewolucyjne algorytmy potwierdzają neodarwinistyczny mechanizm, jakoby dobór naturalny i przypadkowe mutację były główną kreacyjną siłą w biologii.
Jednakże dokładna analiza ewolucyjnych algorytmów i informacji, za pomocą której programuje się je pokazuje, że ewolucyjne algorytmy stwarzają problem, a nie stanowią rozwiązanie dla kwestii pochodzenia informacji i kompleksowości w żywych organizmach. Nie rozwiązując problemu jedynie spychają go głębiej.
Algorytmy ewolucyjne — czym są?
Algorytmy ewolucyjne i genetyczne to ściśle zdefiniowane matematyczne procedury stosowane do rozwiązywania niektórych problemów optymalizacyjnych (lub decyzyjnych), dla których standardowe algorytmy są nieznane lub mają nieakceptowalnie długi czas działania.
Twierdzi się o nich, że zasada ich działania naśladuje mechanizmy znane ze świata przyrody, tj. doboru naturalnego i dziedziczności. Algorytm genetyczny rozpoczyna pracę przez porównanie licznych początkowych projektów z ustalonym wcześniej zespołem kryteriów oceny. Projekty najbardziej pasujące do tych kryteriów są selekcjonowane i poddawane niewielkim losowym zmianom — "mutacjom" i "reprodukcji". W wyniku "krzyżowania się" pochodzącej od "rodziców" informacji, jak i przypadkowym "mutacjom" powstają nowe warianty projektów, które ponownie oceniane są przez ustalone kryteria, a te najlepsze selekcjonowane są do dalszej "ewolucji". Genetyczny algorytm powtarza ten schemat tak długo, aż pojawi się optymalny projekt, to jest najlepiej spełniający założone wcześniej kryteria oceny. Mamy tu więc do czynienia ze swoistym procesem optymalizacji. Algorytmy ewolucyjne zawierają w sobie element losowości konieczny do otrzymania najlepszych wyników optymalizacji. To powoduje, że są one często przedstawiane jako doskonała ilustracja, a nawet potwierdzenie, teorii biologicznej ewolucji. Jest to jednak twierdzenie fałszywe.
Aby algorytmy ewolucyjne znalazły rozwiązanie jakiegoś problemu, konieczna do tego informacja musi zostać do nich rozumnie wprowadzona w samej konstrukcji takiego algorytmu, jak i w specjalnej funkcji, która ocenia wyniki (najczęściej jest to tzw. funkcja dostosowania lub oceny — fitness function). Każdy więc projekt, jaki generują algorytmy ewolucyjne wymaga najpierw wprowadzenia projektu — w konstrukcji takiego algorytmu i w informacji, która steruje takim algorytmem. Naukowcy badający ewolucyjne algorytmy realizują tym samym zasadę, że aby ewolucyjny algorytm wytworzył jakiś projekt wymaga najpierw wprowadzenia do niego projektu. Algorytmy, o których twierdzi się, że potwierdzają niewyobrażalne kreacyjne moce przypadku, spełniać muszą m.in. następujące warunki:
- Musi powstać projekt poszukiwania jakiegoś rozwiązania.
- Muszą zostać precyzyjnie zdefiniowane kryteria oceny (selekcji) testowanych wyników pod kątem dobranego wcześniej celu.
- Musi powstać odpowiednie oprogramowanie konieczne dla ewolucyjnego algorytmu.
- Choć przypadkowe impulsy grają pewną rolę w odnajdywaniu optymalnego rozwiązania, są one tylko niewielką częścią precyzyjnie zaprojektowanej struktury, która jest konieczna by to rozwiązanie zostało odnalezione. Same przypadkowe impulsy nigdy niczego nie zaprojektują, jeśli nie będą tylko niewielką częścią ściśle deterministycznego procesu.
- Nawet te przypadkowe impulsy generowane są przez specjalny do tego celu skonstruowany algorytm — generator liczb pseudolosowych.
Pod tym względem bardzo pouczające są słowa Geoffrey’a Millera z University College w Londynie:
Algorytmy genetyczne sprawdzają się przy poszukiwaniu raczej prostych rozwiązań w małej przestrzeni projektów. Jednak dla trudnych problemów i bardzo dużej przestrzeni projektów, zaprojektowanie dobrego genetycznego algorytmu jest bardzo, bardzo trudne. Cała wiedza jaką nasi inżynierowie mogą wykorzystać stojąc w obliczu projektowania — specjalistyczna wiedza, inżynieryjne zasady, narzędzia analityczne, metody heurystyczne i tym podobne — musi być wbudowana w genetyczny algorytm. [2]
Gdyby na poważnie brać zapewnienia niektórych ewolucjonistów, że algorytmy ewolucyjne są dobrą ilustracją biologicznej ewolucji, znaczy to, że ewolucja ta jest procesem, w którym inteligencja gra ogromną rolę. A więc otrzymalibyśmy formę kreacjonizmu. Wynika z tego, że ewolucyjne algorytmy (i neodarwinistyczny mechanizm w szczególności) są niezdolne do rozwiązania problemu spontanicznego, naturalistycznego powstawania projektów, jakie widzimy np. w żywych organizmach. Nie zważając na to, wielu koryfeuszy ewolucjonizmu bezustannie przywołuje algorytmy ewolucyjne jako analogię procesu biologicznej ewolucji.
Symulacja Dawkinsa
Najbardziej znaną taką demonstracją ewolucji jest program Richarda Dawkinsa zwykle nazywany WEASEL, zaprezentowany w jego książce Ślepy zegarmistrz. W symulacji tej potęga doboru kumulatywnego pokazana jest jako procedura zaczynająca się od przypadkowej sekwencji liter (takich, jakie mogły być napisane np. przez małpę chaotycznie uderzającą w klawiaturę komputera) i stopniowo przekształcającą się w hamletowską sentencję METHINKS IT IS LIKE A WEASEL (Zdaje mi się, że jest podobniejsza do łasicy). [3]
W symulacji tej komputer generuje przypadkowy ciąg liter reprezentujących wyjściowy "organizm". Ta sekwencja zawsze zawiera dokładnie taką samą liczbę liter jak sekwencja docelowa. Ta pierwsza sekwencja jest następnie powielana do 100 kopii — każda "generacja" zawiera właśnie 100 takich sekwencji, czyli "organizmów". Następnie w każdej z tych 100 sekwencji następuje jedna przypadkowa "mutacja" w przypadkowym miejscu, polegająca na zastąpieniu jakiejś litery inną (substytucja). Teraz, co jest rzekomo analogiczne do doboru naturalnego, każda taka sekwencja jest testowana w celu określenia, która jest najbardziej zbliżona do docelowej sekwencji METHINKS IT IS LIKE A WEASEL. Jedna (tylko jedna) z tych 100 sekwencji, która najbardziej przypomina docelowe zdanie jest selekcjonowana do dalszej "ewolucji". Ta najbardziej obiecująca sekwencja jest następnie znowu kopiowana do 100 "osobników". I znowu każda z tych 100 sekwencji podlega przypadkowej "mutacji", a sekwencja najbardziej zbliżona do docelowej jest selekcjonowana, by stała się wyjściową dla kolejnej "generacji" — co ma reprezentować reprodukcję. W tym cyklu powtarza się to, aż ewoluująca sekwencja stanie się sekwencją docelową.
Z prostego rachunku prawdopodobieństwa biorącego pod uwagę: długość sekwencji, liczbę liter w alfabecie, tempo "mutacji" dla każdego "osobnika" i ilość osobników w generacji wynika, że "ewolucja" przypadkowo wygenerowanego ciągu liter zacznie stopniowo coraz bardziej przypominać hamletowską frazę, aż w końcu stanie się z nią identyczna, to jest wszystkie litery i spacje znajdą się na właściwym miejscu (potrzeba na to zwykle mniej niż 100 "generacji").
Symulacja ta ma ilustrować ewolucję przez dobór kumulatywny faworyzujący korzystne mutacje (więcej na temat tej symulacji patrz w Przewodniku po programie Weasel).
Dawkins krytykuje swoją symulację
Analogia Dawkinsa była ostro krytykowana zwłaszcza ze strony społeczności kreacjonistów, którzy argumentowali, że nie może ona być analogią ewolucji, ponieważ z góry określa jej stały cel — w tej symulacji możliwy był tylko jeden wynik, mianowicie osiągnięcie wybranej tekstowej sekwencji; symulacja była tak zaprogramowana, że ten wynik był nieunikniony. Trzeba jednak zauważyć, że pierwszym surowym krytykiem tej symulacji był sam Richard Dawkins. Krytykę tę przedstawił on zaraz po omówieniu swojej symulacji:
Co prawda model małpy/Szekspira dobrze się nadaje do wyjaśnienia różnicy między doborem jednorazowym a doborem kumulatywnym, jednak pod pewnymi ważnymi względami jest mylący. Przede wszystkim — w każdym pokoleniu podlegającym selektywnej reprodukcji zmutowane sekwencje potomne oceniano na podstawie kryterium ich podobieństwa do odległego idealnego wzorca, jakim było zdanie METHINKS IT IS LIKE A WEASEL. W życiu tak nie jest. Ewolucja nie ma długoterminowego celu. Nie ma żadnego odległego wzorca, nic ostatecznie doskonałego, co mogłoby odegrać rolę kryterium doboru (...). [4]
Wynika z tego, że program WEASEL nie był nigdy zamierzony jako demonstracja ewolucji — chociaż był często i niewłaściwie cytowany jakoby nią był. Przykładowo symulację Dawkinsa jako dobrą analogię do ewolucyjnych procesów pod niebiosa wychwala internetowy podęrcznik serwisu Biologia Molekularna w Internecie opracowywany przez studentów IV roku Wydziału Biologii Uniwersytetu Warszawskiego przy wsparciu Fundacji im. Stefana Batorego. Podęrcznik ten w rozdziale pt. "Elementy ewolucji molekularnej" w podrozdziale "Dobór naturalny" stwierdza:
Krytycy teorii doboru powątpiewają, czy z losowych mutacji, które są jedynie błędami przy kopiowaniu informacji genetycznej mogą powstać skomplikowane, a przez to mało prawdopodobne układy.
Na tego rodzaju krytykę, zwolennicy teorii doboru odpowiadają w następujący sposób: każda zmiana w materiale genetycznym, zachodząca z małym prawdopodobieństwem, może pogorszyć lub poprawić dostosowanie. Biorąc pod uwagę fakt, że organizmy się rozmnażają, uzyskujemy wiele różnych układów, z których jedne są lepiej, drugie gorzej przystosowane. Działanie doboru polega na dopuszczaniu do dalszego rozrodu tylko lepiej przystosowanych.
W celu lepszego wyjaśnienia tego zagadnienia można przytoczyć rozumowanie, którym posłużył się Richard Dawkins w książce Ślepy zegarmistrz. Dawkins stara się udowodnić, że dobór naturalny, bazując na przypadkowych układach, jest w stanie doprowadzić do powstania złożoności, którą dostrzegamy w naturze. Rozważane jest tam zdanie, będące cytatem z Hamleta, które brzmi: METHINKS IS LIKE A WEASEL (Zdaje mi się, że jest podobniejsza do łasicy przeł. J. Paszkowski). [Studenci nieco zniekształcili oryginalną sentencję — M.O.] Istnieje bardzo małe prawdopodobieństwo (1 na 1040), aby uzyskać takie zdanie z wszystkimi literami na właściwym miejscu, naciskając przypadkowe klawisze na klawiaturze komputera. Tak samo jak niemożliwe jest uzyskanie skomplikowanych układów w jednym kroku; na drodze tzw. "doboru jednorazowego". Dawkins rozważa jednak inny sposób według, którego może działać dobór, nazywa go "doborem kumulatywnym". Wyobraźmy sobie, że komputer wybiera przypadkową sekwencję 28 znaków (w przypadku Dawkinsa było to WDLMNLT DTJBKWIRZRZELMQCO P), następnie kopiuje w kilkunastu egzemplarzach (replikacja). Ale kopiując owe znaki, przypadkowo z pewnym stałym prawdopodobieństwem robi błędy (mutacje). Następnym krokiem jest wybór spośród uzyskanych sekwencji, tych które są najbardziej podobne do poszukiwanej. W ten sposób symulowane są procesy replikacji z mutacjami oraz selekcji. Oto co udało się uzyskać powtarzając ten zabieg, przez kolejne 40 pokoleń: (pokazano tylko co dziesiąte pokolenie):
pokolenie 10: MDLDMNLS ITJISWHRZREZ MECS P,
pokolenie 20: MELDINLS IT ISWPRKE Z WECSEL,
pokolenie 30: METHINGS IT ISWLIKE B WECSEL,
pokolenie 40: METHINGS IT IS LIKE I WEASEL,
W czterdziestym trzecim pokoleniu udało się ostatecznie uzyskać cel!
Jasne, że udało się osiągnąć cel, ponieważ symulacją Dawkinsa steruje deterministyczna procedura, która zawsze gwarantuje osiągnięcie docelowej sentencji. Można zrozumieć entuzjazm studentów wobec symulacji Dawkinsa jako wynikający z młodzieńczych, gorących emocji. Jednak za pomoc i merytoryczną oceną tekstów tego podręcznika studenci dziękują profesorom: Ewie Bartnik, Andrzejowi Jerzmanowskiemu i Piotrowi Stępniowi. Najwyraźniej wymienionym profesorom również umknął uwadze fakt, że sam Dawkins stwierdził, że jego symulacja nie jest dobrą ilustracją doboru naturalnego. [5]
Nowsze symulacje procesu ewolucji
Są nowsze próby ewolucyjnych symulacji, godne zauważenia są tu symulacja Schneidera EV opisana w Nucleic Acids Research [6], jak również symulacja Lenskiego et al. AVIDA, która została przedstawiona w Nature. [7] W obu tych symulacjach ich autorzy stwierdzają, że nie ma w nich żadnego określonego stałego celu, w rzeczy samej możliwe było uzyskanie wielu różnych wyników i stąd, jak się wydaje, autorom tych symulacji udało się przezwyciężyć zastrzeżenie Dawkinsa do jego własnego modelu, polegające na tym, iż ewolucja nie ma długoterminowego celu.
Wciąż teleologia...
Jednak w obu przypadkach autorzy nie unikają zarzutu Dawkinsa, tylko spychają problem nieco głębiej. Obydwa algorytmy wciąż stale dążą do z góry ustalonego odległego celu ("odległego idealnego wzorca", by użyć słów Dawkinsa), tyle, że z subtelną różnicą. Ów cel nie jest genomem pojawiającym się na końcu procedury symulacyjnej, lecz wynikiem zastosowania algorytmu przetwarzającego do genomu końcowego.
W przypadku symulacji Schneidera cel jest z góry określonym układem ustalonych bitów, które wskazują lokalizacje "połączeń" (binding sites) symulowanego "stworzenia" opartego na DNA. Genom jest przetwarzany tak, by pierwszych 125 miejsc zostało zakodowane jako "macierz wag" (matrix weight). Ta macierz jest następnie używana do przetworzenia reszty genomu w celu ustalenia, które miejsca zostaną "rozpoznane" jako połączenia, a które nie. Przeżycie genomu zależy od tego, na ile wynik działania wspomnianego algorytmu pasuje do założonego z góry i ustalonego układu miejsc odpowiadających połączeniom. Wiele możliwych układów genetycznych spełnia kryteria celu, zatem funkcja dająca wynik jest wielo-jednoznaczna.
Innymi słowy w symulacji EV algorytm ze zdefiniowaną funkcją oceny skanuje ewoluujący genom dając wyniki w formie ciągu symboli składających się z dwóch stanów — "prawda" i "fałsz". Przeżycie organizmu jest determinowane przez porównanie tych właśnie wyników skanowania z ciągiem symboli docelowych, które pełnią rolę wzorca. W przypadku Schneidera taką docelową sekwencją symboli jest ciąg zmiennych typu boolowskiego (boolean) — "prawda" i "fałsz".
Tak, jak symulacja Dawkinsa, symulacja Schneidera startuje z losowo wybranego "genomu" i nie wymaga dalszej interwencji. Inaczej jednak niż w symulacji Dawkinsa, w tej Schneidera nie jest wyraźnie zdefiniowana docelowa sekwencja. Mimo tego, symulacja ta definiuje docelową sekwencję pośrednio przez wybór funkcji dostosowania (fitness function). Schneider musiał rzecz jasna zaprogramować w swoim algorytmie dowolne określenie liczby pomyłek. Określenie liczby pomyłek jest kluczową cechą definiującą jego funkcję dostosowania. Tu optymalne dostosowanie odpowiada minimalnej liczbie błędów. Co więcej, przez powiązanie dostosowania z liczbą błędów, Schneider zagwarantował, że gradient jego funkcji dostosowania stopniowo wzrasta i w ten sposób jego ewolucyjny algorytm dąży w krótkim czasie do optymalnej sekwencji (kryteria optymalności zostały wcześniej zdefiniowane w relacji do funkcji dostosowania).
Ta symulacja nie pokazuje więc ewolucji informacji od — wedle słów Schneidera — "kompletnie przypadkowego genomu" bez żadnej dodatkowej interwencji, ponieważ jest rodzajem "sterowanej ewolucji" w formie prostej sieci neuronowej (tzw. perceptron) i posiada stały, określony wcześniej cel, w symulacji Schneidera zawarty w ciągu tzw. sitelocations. Informacja zawarta w tym ciągu składająca się z 16 liczb jest dokładnie równa informacji, która jest generowana w wyniku "ewolucji od zera". Krótko mówiąc stały cel symulowanej ewolucji zawiera całą informację, która kiedykolwiek może się pojawić w wyniku takiej ewolucji.
Oznacza to, że symulacja ta nadziewa się na ten sam krytycyzm, który Dawkins wyraził w stosunku do swojej symulacji WEASEL, mianowicie, że ten model wymaga określenia długoterminowego celu ewolucji, który w biologicznej ewolucji — jeśli wierzyć ewolucjonistom — nie istnieje.
Z kolei w symulacji Lenskiego et al. zastosowane są cyfrowe "organizmy" — komputerowe programy, które potrafią się replikować, są obiektem mutacji i rywalizują ze sobą. Jej autorzy przechwalają się, że ich symulacja pokazuje "jak złożone funkcje mogą powstawać przez przypadkowe mutacje i dobór naturalny". [8] W symulacji tej ewoluuje ciąg liter (1 litera z 26). Potem każdy z 26 możliwych kodów traktuje się jako instrukcję dla "wirtualnej maszyny" pracującej w języku zwanym AVIDA. Program ma wygenerować pewne funkcje logiczne (najbardziej złożona z nich jest EQU, czyli test tożsamości dwóch 32-bitowych wyników). Nagradza się (nagrody mają niejednakową wartość) złożoność wyliczonej funkcji, przy czym EQU jako najbardziej skomplikowana jest nagradzana najwyżej. Autorzy tej symulacji triumfalnie obwieszczają:
Nasze eksperymenty pokazują słuszność hipotez sformułowanych po raz pierwszy przez Darwina i popartych dzisiaj przez porównawcze i eksperymentalne świadectwo, że złożone cechy generalnie ewoluują przez modyfikacje istniejących struktur i funkcji. [9]
Ich przechwałki są jednak bez pokrycia. Symulacji AVIDA można postawić dokładnie ten sam zarzut, który Dawkins postawił w stosunku do swojej symulacji WEASEL — założenia długoterminowego celu ewolucji. Aby określić prawdopodobieństwo przeżycia cyfrowego organizmu testuje się każdy program przy pomocy zadanego zbioru 32-bitowych wartości wejściowych, a potem 32-bitowe wyniki porównuje się z ustalonym zbiorem wymaganych wyników (tj. funkcja EQU musi zostać zawczasu wyliczona, potem używa się jej jako celu). Funkcja EQU jest więc docelowym wzorcem sterującym ewolucją. Faktycznie, aby odpowiednio ukierunkować ewolucję, trzeba było zrobić znacznie więcej, przedzielając nagrody pośrednie za łatwiejsze do obliczenia funkcje logiczne. [10]
Podsumowując, można stwierdzić, iż tak EV, jak i AVIDA wymagają wyznaczenia jakiegoś celu przed rozpoczęciem biegu ewolucji, a ów cel powinien być wynikiem zastosowania pewnego algorytmu komputerowego do genomu. Obie procedury ogłoszono "pozbawionymi celu" w tym sensie, że dają wiele możliwych wyników spełniających kryteria celu, ale w rzeczywistości obie są związane z ustalonymi wcześniej celami. Tym samym nie mogą stanowić one analogii biologicznej ewolucji, ponieważ, jak wyjaśnia Richard Dawkins:
"Zegarmistrz" — czyli kumulatywny dobór naturalny — jest ślepy i nie widzi w odległej przyszłości żadnego celu. [11]
Nic za darmo
Dokładniejsza analiza algorytmów ewolucyjnych precyzyjnie demonstruje dlaczego nie mogą one służyć jako analogia (czy nawet potwierdzenie — jak chce Lenski ze współpracownikami) naturalistycznej biologicznej ewolucji: istnienie i funkcjonowanie mechanizmu bazującego na próbach i błędach wymaga wcześniejszego zaprojektowania wielu fundamentalnych parametrów (jak zdefiniowanie długoterminowego celu ewolucji i określenie kryteriów selekcji wyników), które same są poza zasięgiem tego mechanizmu. Parametry te muszą zostać określone zanim mechanizm prób i błędów zacznie operować, determinują one całkowicie teren, na którym ten mechanizm może operować, określają jakie cechy będą selekcjonowane i nawigują cyfrową ewolucję w kierunku żądanej optymalizacji. To ten "informacyjny kontekst" wprowadzony do ewolucyjnego algorytmu przede wszystkim odpowiada za skuteczność tych algorytmów. Jest to proces nierozerwalnie związany z celowością. Według Henry‘ego Quastlera, biologa i fizyka, jednego z pierwszych, który w naukach biologicznych zastosował zaawansowane idee teorii informacji:
Stworzenie nowej informacji jest zwykle związane ze świadomą aktywnością. [12]
Algorytmy ewolucyjne odnoszą sukces tylko wtedy, gdy odpowiednio — przez wybór właściwej funkcji dostosowania — sterujemy poszukiwaniami w kierunku funkcji docelowej. W programach tych selekcyjnym kryterium jest przyszła, a nie aktualna funkcja. Taka dalekowzroczna, nawigowana selekcja nie ma odpowiednika w biologicznej ewolucji. W świecie biologicznym, gdy przeżycie organizmów zależy od utrzymania istniejących funkcji, dobór naturalny nie może selekcjonować nowej funkcji, zanim ona nie powstanie. Drugim warunkiem takiej ewolucji jest to, że ewoluująca funkcja musi zachować cały czas swoją funkcjonalność, w przeciwnym wypadku będzie zmniejszała dostosowanie danego organizmu, wskutek czego taki organizm nie będzie miał szans przejść przez sito doboru naturalnego. Poniższa grafika ilustruje ten problem.
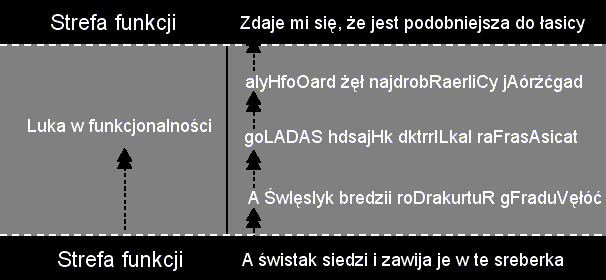 |
Ilustracja ta unaocznia ewolucyjny problem, który symulacja Dawkinsa i inne na niej oparte kompletnie ignoruje. Symulacja ta faworyzuje i selekcjonuje każdy krok w kierunku docelowej sekwencji, nie zawracając sobie głowy niefunkcjonalnymi "formami pośrednimi". Sekwencja znaków zanim zyska nowe znaczenie, musi najpierw stracić znaczenie pierwotne i przebyć szereg niefunkcjonalnych stadiów. Osiąga ona cel tylko dlatego, że selekcyjnym kryterium jest przyszła funkcja. Przykładowo w symulacji Dawkinsa pierwsze w miarę zrozumiałe angielskie wyrazy pojawiają się najwcześniej po 10-15 "generacjach" (iteracjach). W naturze jest to oczywiście nierealne. Dobór naturalny działa na obecne, a nie na przyszłe (a więc nieistniejące) funkcje. Każde pogorszenie się funkcjonalności, nawet jeśli w dalszej perspektywie wiedzie do nowej funkcjonalności, będzie przez dobór naturalny eliminowane.
To czego brakuje doborowi naturalnemu, nie brakuje natomiast doborowi sterowanemu inteligencją, który może wyznaczać długodystansowe cele. Rozumnie można wyznaczyć kryteria selekcji zanim funkcjonalne struktury zaistnieją — tak jak to robi symulacja Dawkinsa i inne algorytmy ewolucyjne. Takie algorytmy nie są więc tylko "skrajnie uproszczonymi" modelami ewolucyjnych procesów, jak tłumaczą niektórzy ewolucjoniści. Ich sukces zależy od wcześniejszego wyznaczenia celu(ów) ewolucji i określenia kryteriów selekcji, które mogą prowadzić w kierunku tego celu(ów), niezależnie od tego, czy są to cele stałe czy zmienne. Czyli ich sukces zależy od tego, czego w biologicznej ewolucji właśnie nie ma.
Jeśli więc algorytmy ewolucyjne są w miarę dobrym odwzorowaniem rzeczywistych biologicznych procesów, znaczy to, że proces ewolucji jest zależny od ustalonych wcześniej celów i w ten sposób sterowany. Problem w tym, że taka idea nie wzbudziłaby zachwytu ewolucjonistów.
Zasadniczą sprawą, którą trzeba podkreślić jest ta, że rozmaite "symulacje ewolucji" od prościutkich programów w rodzaju WEASEL-a Dawkinsa po bardziej wyrafinowane, jak AVIDA są intencjonalnie napisane w celu osiągnięcia żądanego wyniku. To zresztą typowe ewolucjonistyczne rozumowanie: startujemy z wnioskiem, że "ewolucja jest prawdziwa", następnie "dowodzimy" ten wniosek robiąc symulację z góry napisaną tak, aby przesądzała ona wynik, po czym ogłaszamy to, jako kolejne wspaniałe potwierdzenie prawdziwości teorii ewolucji.
Amerykański matematyk, Williama Dembskiego omawiając ewolucyjne algorytmy stwierdza krótko: "nic za darmo" — informacja generowana przez ewolucyjny algorytm nie jest generowana z niczego, ale wymaga wcześniejszego wprowadzenia takiej informacji do tego algorytmu. Algorytm ewolucyjny tylko przetwarza (m.in. przez zmienność i selekcję) już obecną w nim informację. [13]
Przykładem może być tu poszukiwanie optymalnego kształtu anteny nadającej równomiernie we wszystkich kierunkach ponad krzywizną ziemi. W przeciwieństwie do przewidywań, symetryczne anteny o geometrycznych kształtach nie rozwiązywały tego problemu optymalnie. Inżynierowie Edward Altshuler i Derek Linden zastosowali więc w poszukiwaniu projektu takiej anteny algorytm genetyczny. Najlepszą okazała się antena wyglądająca na śmieszną plątaninię drutów (crooked wire genetic antennas). Co więcej, znaczące jest, że genetyczny algorytm wyszukał ją wśród innych podobnie fantazyjnie poskręcanych anten, z których większość jednak nie pracowała właściwie. [14]
Zanim jednak Altshuler i Linden napisali genetyczny algorytm do znajdowania takich anten, byli oni po prostu zainteresowani w ich znalezieniu. Dokładnie zdefiniowali co szukają i jakie kryteria musi spełniać przyszła antena. Stworzona przez nich funkcja dostosowania mierzyła stopień w jakim kolejne testowane anteny nadawały równomiernie. Funkcja ta nie spadła z nieba, ale była precyzyjnie zaprojektowana na bazie ich naukowej wiedzy. Od momentu gdy inżynierowie ci zdefiniowali właściwą funkcję dostosowania, rozwiązali tym samym oryginalny problem. Pozostało tylko, by algorytm genetyczny testował pod kątem zdefiniowanych wcześniej kryteriów różne losowo "powykrzywiane" anteny. I odnalazł tę, która najlepiej spełnia zadane kryteria. [15]
Analogicznie było w przypadku projektu anteny do satelitów wykonanej przez specjalistów z NASA. Opracowali oni oprogramowanie działające w sieci 120 komputerów osobistych, a wykorzystujące algorytm genetyczny do znalezienia optymalnego projektu anteny. Program testował pod kątem dobranych wcześniej kryteriów miliony potencjalnych anten, by wybrać tę optymalną. Jason Lohn z NASA Ames Research Center położonego w kalifornijskiej Krzemowej Dolinie wyjaśnia:
Powiedzieliśmy programowi jakie właściwości powinna mieć antena, a komputer symulował ewolucję, zachowując najlepsze projekty anten, które spełniały kryteria, o jakie pytaliśmy. [16]
Tutaj podobnie sukces był możliwy tylko dlatego, że funkcja oceny testowała różne projekty anten pod kątem przyszłego celu, tak aby jak wyjaśnia Lohn rezultat "spełniał pożądane specyfikacje".
Krótko mówiąc, koniecznym warunkiem, aby algorytm ewolucyjny znalazł optymalne rozwiązanie problemu jest takie zdefiniowane kryteriów selekcji, aby operowały one pod kątem przyszłego celu. Potwierdza to Melanie Mitchell:
Początkowy wybór stałego systemu oprogramowania sterującego genetycznym algorytmem stanowi paradoks dla potencjalnego użytkownika takiego algorytmu: dla każdego problemu, którego rozwiązanie jest dostępne dla genetycznego algorytmu, użytkownik nie wie jak rozwiązać taki problem dopóki, dopóty nie zaprojektuje dla tego algorytmu właściwej procedury poszukującej rozwiązania. W rzeczywistości stworzenie takiej właściwej procedury jest zawsze równoznaczne z rozwiązaniem oryginalnego problemu! [17]
Reasumując, algorytmy ewolucyjne i genetyczne nie mogą być dobrym odwzorowaniem ewolucyjnych procesów z dwóch zasadniczych powodów:
- Kryteria selekcji operują pod kątem przyszłej, a nie aktualnej funkcji. To znaczy, że musi zostać predeterminowany odległy cel lub cele takiej ewolucji.
- Informacja konieczna do wytworzenia jakiegoś projektu lub złożoności musi zostać wprowadzona do ewolucyjnego algorytmu zanim zacznie on działać. Ta informacja pozwala osiągnąć optymalizację pod kątem dobranego wcześniej celu.
Żaden z tych warunków w biologicznej ewolucji nie występuje. Z powyższego wynika, że algorytmy ewolucyjne są przykładem projektu, w którym element przypadkowości stanowi część znacznie większego deterministycznego procesu i został "zaprzęgnięty" do inteligentnego projektu — optymalizacji pewnych rozwiązań.
Jeśli chcesz wiedzieć więcej:
William A. Dembski, Why Natural Selection Can't Design Anything. Doskonała i przystępnie napisana analiza amerykańskiego matematyka gładko dokumentująca dlaczego algorytmy ewolucyjne nie mogą rozwiązać problemu pochodzenia informacji i projektów w żywych organizmach. (Do przeczytania artykułu konieczny jest Adobe Acrobat Reader)
Royal Truman, The Problem of Information for the Theory of Evolution. Świetna techniczna krytyka symulacji EV Schneidera autorstwa niemieckiego informatyka i chemika.
Darel R. Finley, Three Issues With No Free Lunch. Przystępne streszczenie technicznych argumentów Williama Dembskiego z jego książki No Free Lunch dotyczących algorytmów ewolucyjnych, w której precyzyjnie udokumentował, że algorytmy ewolucyjne nie rozwiązują problemu pochodzenia projektów w żywych organizmach. (Do przeczytania artykułu konieczny jest Adobe Acrobat Reader)
Karl D. Stephan, What Does Evolutionary Computing Say About Intelligent Design? Przystępne wyjaśnienie dlaczego algorytmy ewolucyjne są argumentem za rozumnym projektem, a nie za naturalistyczną ewolucją. (Do przeczytania artykułu konieczny jest Adobe Acrobat Reader)
David Owen, A Shot in the Dark. Solidna analiza metod działania ewolucyjnych algorytmów. (Do przeczytania artykułu konieczny jest Adobe Acrobat Reader)
I.G.D. Strachan, An Evaluation of "Ev". Techniczna analiza wspomnianej wyżej symulacji Schneidera pokazująca, że nie udało się jej uniknąć zarzutu Dawkinsa o długoterminowym celu ewolucji. (Do przeczytania artykułu konieczny jest Adobe Acrobat Reader)
Royal Truman, Dawkins’ weasel revisited. Matematyczna analiza słynnej symulacji Dawkinsa potwierdzająca, że jest ona determinowana do osiągnięcia docelowej sekwencji.
John R. Bracht, Inventions, Algorithms, and Biological Design. Doskonały przegląd dokumentujący, że algorytmy genetyczne dowodzą, że darwinistyczny mechanizm nie jest w stanie stworzyć niczego nowego. (Do przeczytania artykułu konieczny jest Adobe Acrobat Reader)
Royal Truman, Evaluation of neo-Darwinian Theory with Avida Simulations. Krytyczna analiza symulacji AVIDA. Poziom techniczny. (Do przeczytania artykułu konieczny jest Adobe Acrobat Reader)
Don Batten, Genetic Algorithms. Do They Show that Evolution Works? Przystępne wyjaśnienie dlaczego ewolucyjne algorytmy nie mogą stanowić dobrej analogii dla biologicznej ewolucji.
Inne algorytmy ewolucyjne do ściągnięcia:
Mutation program. (Do uruchomienia programu wymagany jest Microsoft Access 97, 2000 lub XP) Świetny i przejrzysty algorytm genetyczny. Posiada wiele dodatkowych funkcji np.: substytucje, insercje, delecje, różna częstotliwość mutacji w różnych częściach genomu itp.
MESA: Monotonic Evolutionary Simulation Algorithm . Algorytm MESA modeluje ewolucyjne przeszukiwanie przez użycie stałego gradientu fitness. To implikuje, że krajobraz dostosowania stopniowo dąży do pojedynczego optimum (szczytu lub doliny) i bada jak szybko ewolucja może znajdować lokalne optimum kiedy fitness jest przypadkowo zaburzana. Ten fachowy algorytm zawiera liczne zmienne, które można modyfikować. Do jego uruchomienia wymagany jest Java Runtime Environment (JRE) — linki do pobrania znajdują się na stronach MESA.
Michał Ostrowski
Przypisy
[1] Zob. John H. Holland, Adaptation in Natural and Artificial Systems, University of Michigan Press, Ann Arbor, MI 1975 (Massachusetts Institute of Technology Press 2nd edition 1992). Zob. także John H. Holland, Genetic algorithms, Scientific American 1992, vol. 267, No. 1, s. 44-50.
Niezależnie od Hollanda podobnie działające algorytmy rozwijali w Zachodnich Niemczech inżynierowie Ingo Rechenberg i Hans-Paul Schwefel (por. Ingo Rechenberg, Evolutionsstrategie: Optimierung technischer Systeme nach Prinzipien der biologischen Evolution, Frommann-Holzboog, Stuttgart 1973; Hans-Paul Schwefel, Numerische Optimierung von Computermodellen mittels der Evolutionsstrategie, Birkhauser, Basel 1977).
[2] Geoffrey Miller, Technological Evolution as Self-Fulfilling Prophecy, [w:] John Ziman (ed.), Technological Innovation as an Evolutionary Process, Cambridge University Press, Cambridge 2000, s. 208, podkreślenie dodane.
[3] William Shakespeare, Hamlet, Prince of Denmark, Act III Scene II, [w:] William James Craig, (ed.) The Oxford Shakespeare: the complete works of William Shakespeare, Oxford University Press, London 1914, Bartleby.com, 2000.
[4] Richard Dawkins, Ślepy zegarmistrz czyli, jak ewolucja dowodzi, że świat nie został zaplanowany, Biblioteka Myśli Współczesnej, Państwowy Instytut Wydawniczy, Warszawa 1994, s. 92, podkreślenie dodane.
[5] Takich kwiatków jest więcej. Zob. np. The Weasel Applet — dostępną online w formie apletu symulację odtwarzającą tę Dawkinsa. W komentarzu do niej czytamy m.in.:
Zdolność tego apletu do osiągania zadanych fraz, nie dowodzi oczywiście, że ewolucja miała miejsce. (...) Ale powinniście pomyśleć o tym aplecie, kiedy usłyszycie od kogoś, że jest zbyt nieprawdopodobne by "ślepy przypadek" mógł wytworzyć takie kompleksy, jak — powiedzmy — molekuła hemoglobiny. Kiedy rezultaty są obiektem kierunkowej presji, "ślepy przypadek" może zdziałać o wiele więcej niż sugeruje to wasza intuicja.
Jeśli sami chcecie się przekonać o "genialności" i inwencji tego apletu (i przy tym jesteście nieco złośliwi) zróbcie następujący test: wpiszcie w docelowej sekwencji tego apletu (Goal Phrase) zdanie zawierające polskie znaki (np.: ą, ę, ć, ś itd.), lub jakiś znak specjalny (np. $, &) i zobaczcie co się stanie. Jak łatwo zauważyć, program może "ewoluować" do końca świata, a nie dojdzie do docelowej sekwencji z takimi znakami. Dzieje się tak, ponieważ programista tej symulacji nie zablokował możliwości wprowadzania znaków tego typu w docelowej frazie i nie uwzględnił tego w losowym doborze liter alfabetu, czyli "mutacjach". Znaczy to, że litery losowane są tylko z zakresu alfabetu angielskiego, przez co ewolucja sekwencji zawierającej choćby jeden polski znak czy znak specjalny nie dojdzie nigdy do celu. To pouczający przykład pokazujący, że "ewolucja złożoności" nie stwarza niczego nowego, ona może tylko odtwarzać to, co wcześniej w nią się rozumnie wprowadzi. Wbrew zapewnieniom autora tego apletu, "ślepy przypadek" nie jest w stanie stworzyć nawet jednego znaku specjalnego, nie mówiąc już o molekule hemoglobiny.
[6] Thomas D. Schneider, Evolution of biological information, Nucleic Acids Research 2000, vol. 28, s. 2794-2799.
[7] Richard E. Lenski, Charles Ofria, Robert T. Pennock and Christoph Adami, The evolutionary origin of complex features (pdf, 421 kb), Nature 8 May 2003, vol. 423, s. 139-144.
[8] Lenski et al., The evolutionary origin..., s. 139.
[9] Lenski et al., The evolutionary origin..., s. 143.
[10] Osobnym problemem jest modelowanie darwinowskich procesów w odniesieniu do "genomów" cyfrowych "organizmów". Komputerowe instrukcje nie są odpowiednikami kodonów DNA. Jeśli "cyfrowe organizmy" mają być dobrymi modelami żywych organizmów, należy zrównać poziom kodu źródłowego — faktyczne instrukcje, w których mogą zachodzić losowe mutacje i na które działa dobór naturalny — z kodonami. Ktoś, kto ma nawet niewielkie pojęcie o programowaniu bez problemu odgadnie, co stanie się w przypadku pojawienia się "przypadkowych punktowych mutacji" w kodzie źródłowym sterującym zachowaniem się "cyfrowych organizmów".
[11] Dawkins, Ślepy zegarmistrz..., s. 92.
[12] Henry Quastler, The Emergence of Biological Organization, Yale University Press, New Haven, Connecticut 1964, s. 16.
[13] Por. William A. Dembski, No Free Lunch. Why Specified Complexity Cannot Be Purchased Without Intelligence, Rowman and Littlefield Publishers 2001, s. 183.
[14] Por. Edward E. Altshuler and Derek S. Linden, Design of Wire Antennas Using Genetic Algorithms, [w:] Yahya Rahmat-Samii and Eric Michielssen (eds.), Electromagnetic Optimization by Genetic Algorithms, J. Wiley & Sons, New York 1999, s. 211-248.
[15] Ponieważ przestrzeń wszystkich możliwych konfiguracji drutów składających się na antenę jest ogromna, najlepiej w jej przeszukiwaniu sprawdziło się losowe dobieranie różnych konfiguracji drutów, które następnie były testowane przez funkcję dostosowania (oceny).
[16] John Bluck, Nasa 'Evolutionary' Software Automatically Designs Antenna, Ames News 14 June 2004.
[17] Melanie Mitchell, An Introduction to Genetic Algorithms, Massachusetts Institute of Technology Press, Cambridge, Massachusetts 1998, s. 158, podkreślenie dodane.